![[PukiWiki] [PukiWiki]](image/pukiwiki.png)
![[PukiWiki] [PukiWiki]](image/pukiwiki.png)
前回はプログラミングの基本について説明しました。もう皆さんは、繰り返しと代入と条件分岐については大体イメージがつかめたと思います。まずはとりあえず、ごく簡単なおさらいをやっておきましょう。
次のそれぞれのプログラムの_部分に何を入れるべきかを選択肢から選びなさい。また、Rを使って実行してみましょう。
Rでプログラミングを行う場合は、Rについているスクリプトエディター(R editor)を用いると、操作がずーっと楽になります。
R Editor というエディタのウィンドウが開く
このウィンドウの中に授業ページのサンプルプログラムをペーストして編集
選択した部分のプログラムが実行され、結果がRコンソールに表示される。
for(i in 1:__){ #{と}の間を10回繰り返す。i の値は毎回1ずつ増える
print(i)
}
選択肢: a) 1 b) 5 c) 10
goukei=0 #goukeiという変数に初期値0を代入
for(i in 1:10) {
goukei=_____+i #goukeiに前回までのgoukeiの値にiの値を足したもの代入
}
print(goukei)
選択肢: a) i b) goukei c) print
goukei=0 #goukeiという変数に初期値0を代入
for(i in 1:10) {
if(i %% 2 __ 0) { # %% は割り算をした余りを返す演算子。3 %% 2 の計算結果は 1 になる
goukei=goukei+i # if文の内容は: 「もし i を 2 で割った余りが 0 ならば、{}内を実行
} #それ以外は何もしない
} #for 文のカッコ閉じるに注意
print(goukei)
選択肢: a) = b) == c) <=
この3つの命令それぞれは単純なことなのですが、組み合わせれば、かなり複雑なことでも表現できます。そこで、今回は、上の命令を組み合わせて、シミュレーションに挑戦してみましょう。
右の図は見たことはありますか?これは、遺伝的浮動のシミュレーションの結果で、集団サイズN=100, 対立遺伝子が2つのとき、片方の対立遺伝子の頻度の変化を表したものです。。最初の世代の対立遺伝子の数は50:50。このグラフを使って、遺伝的浮動とはどういうものか、説明できる人はいますか?また、このグラフはどうやって作ったものですか?
プログラミングを知る前は、右のような図を作るのはとうてい無理と思ってたかもしれません。でも、Rを使って、簡単なプログラムを作れば簡単に描けてしまいます。
「生物学科の皆さんになじみが深い」 「Rをつかってやってみて、結果の見た目が面白い」 「思ったよりはプログラミングが簡単」
という3点がこのテーマを選んだ理由です。
なお、シミュレーション(simulation, 「シュミレーションでは無いですよ!」)は、数学モデルなどを用いて現実に似た状況をコンピュータ上で再現することです。模擬実験とも呼ばれています。(大量の計算を必要とするので、Rのようなインタプリタ型のソフトでは、大規模なシミュレーションは難しいです)
これからシミュレーションを行うのですが、つぎのような状況を想定してみましょう。受験勉強でやった集団遺伝のお話を、ちょっとだけ思い出してください。
まず、対象とする架空の生物(ここでは「モナー」を使いました。(注:モナーについて詳しく知りたい人は、ウィキペディアで「モナー」を検索してみてください。))は2倍体の生物で、雌雄の区別が無く、繁殖期になると集団内の個体が一斉に配偶子を非常にたくさん(数千億個!)ずつ放出した後死亡します。配偶子は生息域の空間内をランダムに漂った後、24時間後に、最も近くにある配偶子と接合し、新しい繁殖個体になります。集団の生息域は空間的に限られているため、親世代と同数の子供個体しか、成熟個体になれません。どの子供が成熟個体になるかは、ランダムに(偶然に)決まるものとします。つまり、このモデルだと、世代間で集団サイズに変動は無いということです。モナーにはトンガリ耳と丸耳の2タイプが対立遺伝子(A, a)で決まることが知られています。トンガリ耳の遺伝子型は(AA, Aa)、丸耳の遺伝子型は(aa)です。
なお、上のモナーの説明は、私が今回のシミュレーションのために、適当に設定したものです。簡単に言うと、「任意交配をする有限サイズの生物集団から遺伝子をランダム抽出して、同サイズの次世代集団を作る」ということです
モナー10個体からなる、1つの集団を考えてみましょう。いまこの集団で、対立遺伝子Aの頻度が0.5, 対立遺伝子aの頻度が0.5のとき(10個体だから遺伝子の総数は、20個です。そのうち半分の10個がAで、10個がaです)。集団中の配偶子の接合はランダムに起こると仮定して、100世代の間に、集団中の対立遺伝子の頻度はどのように変化するでしょうか?
今から実験をするのですから、前もって予想しておいた方が面白そうです この集団の遺伝子頻度が1か0に固定するまでに、何世代ぐらいかかると思いますか? 0-25 26-50 51-75 76-100 100<
それでは、次のプログラムを走らせて、20個の遺伝子中対立遺伝子aが10個ある任意交配集団の、100世代の間のaの遺伝子頻度の変化をみてみましょう。
Rでは変数の名前が日本語でも動作します。今回は皆さんの理解を助けるために、変数の値を日本語で書いてみます。プログラムが見づらくなるので、変数名に英文字を使ったものも示しておきました。
まずは、下のプログラムをコピーして、__のところを埋めて実行してみてください。
集団サイズ = __ #1集団内の遺伝子の総数を集団サイズと呼ぶ。今回は20。
対立遺伝子aの数 = __ #対立遺伝子aの集団中の個数を10個がaであるとする
繰り返し世代数 = ___ #今回は100世代観察することにする
for(i in 1:繰り返し世代数){ #この行から対応する}までの間を100世代分(「繰り返し世代数」の値)繰り返す
カウンタ=0 #次世代にランダムに残るaの値を カウンタ という変数に入れる。初期化して0にする。
for(j in 1:集団サイズ){ #配偶子プールから1個の配偶子を取り出す試行を集団サイズの数繰り返す
if ( runif(1) < 対立遺伝子aの数/集団サイズ ){ #乱数を一つ発生させ、
#それが対立遺伝子aの頻度よりも小さければ
カウンタ = カウンタ + 1 #aの個数を入れるcountaという変数の値を1つ増やす
}
}
結果=c(i, 対立遺伝子aの数/集団サイズ) #何世代目かを表す数字(i)と対立遺伝子aの頻度を結果というオブジェクトに入れる
print(結果) #outの内容を画面に表示する
対立遺伝子aの数=カウンタ # 対立遺伝子aの数を新しい世代の対立遺伝子aの個数(カウンタの値)で置き換える
}
numa = 10 #対立遺伝子aの集団中の個数を numa という変数で表す。今回は10個がaであるとする
numgen = 100 #何世代について観察するかを numgen で表す。今回は100世代観察することにする
for(i in 1:numgen){ #この行から対応する}までの間を numgen で指定された100世代分繰り返す
counta=0 #次世代にランダムに残るaの値を counta という変数に入れる。初期化して0にする。
for(j in 1:20){ #数千億からなる配偶子プールから1個の配偶子を取り出す試行を20回行う
if ( runif(1) < numa/20 ){ #乱数を一つ発生させ、それが対立遺伝子aの頻度(a/20)よりも小さければ
counta=counta+1 #aの個数を入れるcountaという変数の値を1つ増やす
}
}
out=c(i, counta/20) #何世代目かを表す数字(i)と対立遺伝子aの頻度(a/20)をoutというオブジェクトに入れる
print(out) #outの内容を画面に表示する
numa=counta # aの値を新しい世代の対立遺伝子aの個数(counta)で置き換える
}質問しますので挙手して答えてください。 100世代目が0だった人、 1だった人、 それ以外の数値だった人 固定した世代数 0-10 10 - 20 20-50 51-75 76-100 100<]
上のプログラム、ここまで説明してきた
繰り返し 代入 条件分岐
だけからできていますね。目新しいものと言えば、
runif() 乱数を一つ発生させる。runif(1)とした場合、0から1までの間の値を1つ発生。
だけです。
では、プログラミングの手順を1つずつ、順を追って見てゆきましょう。この過程さえ理解できれば、シミュレーションのプログラムは自分で書けてしまうのです。
なお、プログラミングの作業自体は、「数学の能力」はさほど重要ではないと思います。 私も「数学」は得意じゃないのです。でもプログラミングはある程度はできます。 プログラミングというのは、石頭のコンピュータでも理解できるように手順を分かりやすく論理的に説明するという点で、 むしろ、「国語能力」とか「説明能力」が必要なのだろうと感じています
最初は小さい数で自分の想像が及ぶ範囲の数にしておくと良いと思います。今回は、簡単に、
20個の遺伝子からなる集団 0世代目の対立遺伝子aの数が10(つまり対立遺伝子aの頻度は0.5) この集団を100世代観察する
という条件にしておきます
名前を付けておくと、あとでコンピュータに命令するときに便利だからです。名前は、Rの規則で使えないもの以外なら、何でも良い。自分に分かりやすいようにつける。上で決めた条件で数値が決まっているものは代入しておく。
*自分で考えた変数名と定数
対立遺伝子aの数 = 10 #最初の世代の対立遺伝子aの数。次世代以降は1つ前の世代で抽出されたaの数が代入される
カウンタ = 0 #配偶子プールから、次世代の集団に取り出された対立遺伝子aの数。1世代が終わったあとで、
数字が入るので、初期値として0を入れておく
集団サイズ = 200 #集団の大きさ(つまり、集団に含まれる遺伝子の数)
繰り返し世代数 = 100 #繰り返しの世代数
紙に絵をかいて、「モナー」の生活環を「繰り返し、代入、条件分岐」で説明できるかを考え、言葉にしてみる。
*自分で考えたプログラムの構造。コマンドを使って書かなくても、 とりあえず、構造が理解できるように書いてあればよい。
*100回繰り返す繰り返し文の構造
for(i in 0:100) {
<※ここに繰り返される内容が入る>
}※集団中の遺伝子数分、つまり、20回繰り返す文
for(j in 0:20) {
<※※ここに繰り返される内容が入る>
} ※※の操作:
「配偶子を一つ取り出すとき、遺伝子頻度の確率で、対立遺伝子aをひく。もし、対立遺伝子aを引いたら、
aの数を入れておく変数(カウンタ)の値を1つ増やす」
= 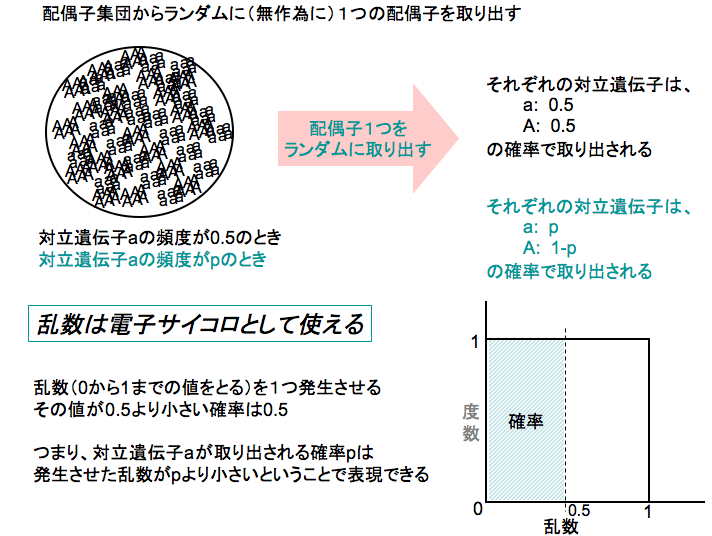
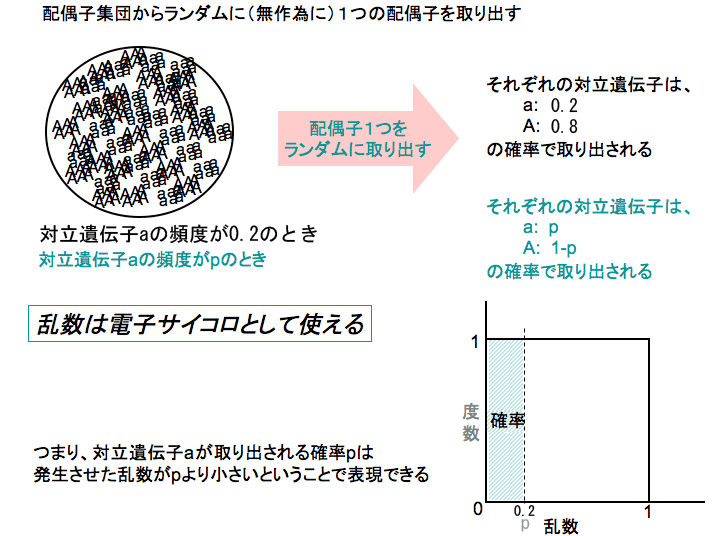
上の『対立遺伝子aを引いたら』(このことは対立遺伝子頻度aの確率で生じる)というのは、
『乱数を一つ発生させたとき、それが対立遺伝子頻度aより低かったら』(これも対立遺伝子頻度aの確率で生じる)と
同じことと考える。そこで、
「乱数を一つ発生させたとき、それが対立遺伝子頻度aより低かったら、
aの数を入れておく変数(counta)の値を1つ増やす」
・内側のfor文の繰り返しが1回終わったら 対立遺伝子aの数 = カウンタ カウンタ= 0 #内側のfor文の直前に書いても同じこと
これでは使いにくいので、aの値を自由に変更できる関数を定義してみましょう。「関数の定義」なんていうと難しそうですが、ようするに、作ったプログラムに名前をつけて、いろいろと数値を変えて解析できるようにすることです。
drift1=function(対立遺伝子aの数){ #関数定義の始まり
繰り返し世代数 = 100 #今回は100世代観察することにする
集団サイズ = 20 #1集団内の遺伝子の総数を集団サイズと呼ぶ。今回は20。
for(i in 1:繰り返し世代数){
カウンタ=0
for(j in 1:集団サイズ){
if ( runif(1) < 対立遺伝子aの数/集団サイズ ){
カウンタ=カウンタ+1
}
}
結果=カウンタ/集団サイズ
print(結果)
対立遺伝子aの数=カウンタ
}
} #関数定義の終わり
さっきのプログラムの上下を関数を定義する関数であるfunction(){}で囲んで、オブジェクトに代入するだけですから、簡単です。この関数を実行するには、最初aの値を()の中に入力して、
drift1(10)
とします。
↑を使って何回も実行してみると、結果がいろいろ変わるのがわかりますよね。
では、せっかく結果が出たのだから、グラフにしてみましょう。でも、その前に、以前使ったplot()という関数を思い出してみましょう。
x=c(1,2,3,4,5) plot(x)
plot()という関数は、ベクトルの内容をプロットしてくれるのでした。そうすると、今のdrift1関数の実行結果はどうやれば表示できるでしょうか?
関数を実行した結果が全て、一つのオブジェクトにベクトルとして格納されていれば良い。例えば: 0.6, 0.55, 0.65, 0.8, 0.95, 1, 1, 1, ...
これをやるには、上のout=count/20としているところを、次から次へとベクトルの要素を付け加えるようにすれば良さそうです。ベクトル要素を付け加える関数が
append()
です。
results=append(results, a/20)
と書けば、resultsというベクトルにa/20の値が追加されます。但し、ひとつだけ重要な注意があります。
resultsというベクトルはあらかじめ存在していなければ、append()は使えない
つまり、for文を使って繰り返しを行う前に、
results=c()
という命令で、空ベクトルを作っておく必要があるのです。
では、まず、結果をresults()に入れてベクトルで返すような変更を、上のプログラムに加えて見ましょう
drift2=function(a){
results=c() #空ベクトルを作る
for(i in 1:30){
counta=0
for(j in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20) #ここにあった out=counta/20 の代わりに、append()関数でベクトル作成
a=counta
}
print(results) #printの文はfor文の外に出したことに注意!理由は分かりますよね
}
これで
[1] 0.50 0.60 0.80 0.70 0.55 0.50 0.35 0.40 0.20 0.30 0.1....
というベクトルが表示できました。これをplot()でグラフにするには、
plot(drift2(10))
とやるだけです。
しかし。。。ちょっと待てよ?どうせなら、関数の中にplot()を入れてしまえばすぐにグラフが表示されるのでは?
drift3=function(a){
results=c() #空ベクトルを作る
for(i in 1:30){
counta=0
for(j in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
plot(results) #ここにあった print文の代わりに、plot()でグラフ表示
}
グラフがすぐに表示できました!でも、点々では見にくいので、線で結んでみましょう。plot()のところを、
plot(results, type="l")
とかえるだけです。
drift4=function(a){
results=c() #空ベクトルを作る
for(i in 1:30){
counta=0
for(j in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
plot(results, type="l") #ここにあった print文の代わりに、plot()でグラフ表示
}
上で作ったプログラムを何度も走らせてみると、グラフの形がどんどん変わります。これはこれで面白いのですが、グラフの軸が一定していないし、1つのグラフに結果が重ねて表示されないので、ちょっと感じがつかめませんね。
そこで、いっそのこと、この実験を20回ぐらい繰り返してみて、結果を1つのグラフに表示させることにしましょう。同じことを何度も繰り返すのですから、for文をもう一つ、外側に作ればいいって、容易に想像がつきますよね?
drift5=function(a){
for(i in 1:20){ #これまでのプログラムの外側に新しく作ったfor文。20回繰り返す。
results=c()
for(j in 1:30){
counta=0
for(k in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
plot(results, type="l")
} #新しく作ったfor文の終わり。ここまで繰り返し。
}
さあ、走らせてみましょう。
drift5(10)
あれ?なんか変ですね。最後は0.0で線が一本でるだけです。
なぜだか分かりますか?
こたえは、aの値です。最初、aは10で与えられましたが、1集団分の繰り返しが終わった後、次の繰り返しの前に、下の数字に戻すのを忘れていました。
drift5=function(a){
firsta=a #関数に渡された(つまり()の中に書かれた)aの値をfirstaというオブジェクトに保存
for(i in 1:20){ #これまでのプログラムの外側に新しく作ったfor文。20回繰り返す。
a=firsta #繰り返しの最初に、aの値を初期値(firsta)に戻す
results=c()
for(j in 1:30){
counta=0
for(k in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
plot(results, type="l")
} #新しく作ったfor文の終わり。ここまで繰り返し。
}
できました。実行してみましょう。ウーン。。。うまくいってるようなのですが、1回ずつ、グラフが変わっちゃいます。グラフを重ねて描くには、par(new=T) というコマンドをplot()の前に使います。
drift5=function(a){
firsta=a #関数に渡された(つまり()の中に書かれた)aの値をfirstaというオブジェクトに保存
for(i in 1:20){ #これまでのプログラムの外側に新しく作ったfor文。20回繰り返す。
a=firsta #繰り返しの最初に、aの値を初期値(firsta)に戻す
results=c()
for(j in 1:30){
counta=0
for(k in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
par(new=T) #複数のグラフを重ね合わせる
plot(results, type="l")
} #新しく作ったfor文の終わり。ここまで繰り返し。
}
あれれ?そうか。始点の軸が固定されていないから、ずれちゃってますね。plot()関数にはylimというオプションがあって、y軸の目盛りを0から1に固定するには、次のように書きます。
drift5=function(a){
firsta=a #関数に渡された(つまり()の中に書かれた)aの値をfirstaというオブジェクトに保存
for(i in 1:20){ #これまでのプログラムの外側に新しく作ったfor文。20回繰り返す。
a=firsta #繰り返しの最初に、aの値を初期値(firsta)に戻す
results=c()
for(j in 1:30){
counta=0
for(k in 1:20){
if ( runif(1) < a/20 ){
counta=counta+1
}
}
results=append(results, a/20)
a=counta
}
par(new=T) #複数のグラフを重ね合わせる
plot(results, type="l", ylim=c(0,1)) #y軸の目盛りを0から1に固定する
} #新しく作ったfor文の終わり。ここまで繰り返し。
}
いよいよ完成にちかづいてきました。図が重なって見にくくなったら、
>frame()
か
>plot.new()
で図を新しく書き直して下さい。
何回かグラフを描かしてみると、わりとすぐに固定してしまうことがわかりますね。じゃあ、もっと、集団サイズがもっと大きかったらどうなるか気になってくるでしょ?
どうすればよいか?プログラムの中で、集団サイズを20に指定しているところを100とかに変更してもいいのですが、どうせなら、関数に値を渡すときに、指定できれば、つまり、
drift(繰り返し数, 世代数, 集団サイズ, 対立遺伝子の数)
とか指定して実行できれば、好きな大きさの集団で、好きな数だけ、シミュレーションを繰り返せますよね。さっきのプログラムで、数値で指定してあったところを変数に置き換えるだけですから、そんなに難しくなさそうです。
では、やってみましょう
drift6=function(x, y, z, a){
plot.new() #ついでに、図を新しく書き直すという関数をここに追加しました
firsta=a
for(i in 1:x){ #上のプログラムでは20でしたが、ここでは、x回繰り返す。
a=firsta
results=c()
for(j in 1:y){ #上のプログラムでは30だったけど、ここではy世代繰り返す。
counta=0
for(k in 1:z){ #上のプログラムでは20だったけど、ここでは集団サイズはz。
if ( runif(1) < a/z ){ #上のプログラムでは20だったけど、ここでは集団サイズはz。
counta=counta+1
}
}
results=append(results, a/z) #上のプログラムでは20だったけど、ここでは集団サイズはz。
a=counta
}
par(new=T)
plot(results, type="l", ylim=c(0,1))
}
}
できちゃいました。 あんまり大きい数字を入れると計算にすごく時間がかかります。途中で終了したいときは、 ESC キーを押してください。
下のプログラムは遺伝的浮動をシミュレートするRのプログラムです。シミュレーションで観察する集団の数(num_repeats)、1集団あたり観察する世代数(num_generations)、集団サイズ(遺伝子数)(size_population)、0世代目における対立遺伝子aの数(num_a_allele)を与えて実行すれば、、ランダムに選ばれる遺伝子が、次世代の集団の遺伝子頻度にどのように影響するかを、シミュレートする集団の数だけグラフに(色つきで)表示することができます。下のプログラムを見て以下の問に答えなさい。
drift= function(num_repeats,num_generations,size_population, num_a_allele){
results=c()
a=num_a_allele
for(i in 1:【1】){
for(j in 1:【2】){
count_a=0
for(k in 1:size_population){
if ( runif(1) < a/【3】 ){
count_a=count_a+1
}
}
a=count_a
results=append(results, a/size_population)
}
a=num_a_allele
}
rmatrix=matrix(results, nrow=num_generations, ncol=num_repeats)
return(matplot(rmatrix, type="l"))
}
num_repeats, num_generations, size_population, num_a_allele
1. 実験の繰り返し数10回、1集団での観察世代数500世代、集団サイズ(遺伝子数)200, 初期状態での対立遺伝子aの数100 2. 実験の繰り返し数10回、1集団での観察世代数100世代、集団サイズ(遺伝子数)20, 初期状態での対立遺伝子aの数10実行するための命令文と、結果のグラフを、1, 2のいずれの場合についても、課題提出ページに貼り付けなさい(注:1番目のシミュレーションの実行には、数分かかるかもしれません)
drift6=function(x, y, z, a){
plot.new() #ついでに、図を新しく書き直すという関数をここに追加しました
firsta=a
for(i in 1:x){ #上のプログラムでは20でしたが、ここでは、x回繰り返す。
a=firsta
results=c()
results_p=c()
for(j in 1:y){
counta=0
count_p=0
count_p_type=0
for(k in 1:z){
if ( runif(1) < a/z ){
counta=counta+1
}
if (k%%2 != 0){ #jが奇数のとき
if ( runif(1) < a/z ){ #もし2つの遺伝子のうち最初のものがaならば
count_p=count_p+1 #count_pの値を1増やす
}
} else { #jが偶数のとき
if ( runif(1) < a/z ){ #もし2つの遺伝子のうち最初のものがaならば
count_p=count_p+1 #count_pの値を1増やす
}
if(count_p == 2){ #連続する奇数回・偶数回で合計したpの値が2ならば(つまり、aが2個だったら)
count_p_type=count_p_type+1 #count_p_typeの値を1増やす(aaとう表現型が1つあると数える)
}
count_p=0 #2個数え終わったら、count_pの値を初期化
}
}
results=append(results, a/z)
results_p=append(results_p, count_p_type/(z/2)) #results_pに集団内のaaの頻度(count_p_type/z/2)を入れる
count_p_type=0 #count_p_typeの値を初期化
a=counta
}
par(new=T)
plot(results, type="l", ylim=c(0,1))
par(new=T)
plot(results_p, type="l", ylim=c(0,1), col="blue") #aaの頻度を青線で表示する
}
}
drift6=function(x, y, z, a, s){ #選択係数sでaaが集団から排除される
plot.new()
firsta=a
for(i in 1:x){
a=firsta
results=c()
results_p=c()
for(j in 1:y){
counta=0
count_p=0
count_p_type=0
for(k in 1:z){
if (k%%2 != 0){ #jが奇数のとき
if ( runif(1) < a/z ){ #もし2つの遺伝子のうち最初のものがaならば
count_p=count_p+1 #count_pの値を1増やす
}
} else { #jが偶数のとき
if ( runif(1) < a/z ){ #もし2つの遺伝子のうち2番目のものがaならば
count_p=count_p+1 #count_pの値を1増やす
}
if(count_p == 2){ #連続する奇数回・偶数回で合計したpの値が2ならば(つまり、aが2個だったら)
if (runif(1) < s) { #遺伝子型がaaのとき乱数を一つとって、それが選択係数より小さければ
k=k-2 #jの値から2を引いて
count_p=0 #count_pを初期化し
next #強制的に次の繰り返し
} else {
count_p_type=count_p_type+1 #count_p_typeの値を1増やす(aaという表現型が1つあると数える)
}
}
counta=counta+count_p
count_p=0
}
}
results=append(results, a/z)
results_p=append(results_p, count_p_type/(z/2)) #results_pに集団内のaaの頻度(count_p_type/z/2)を入れる
count_p_type=0 #count_p_typeの値を初期化
a=counta
}
par(new=T)
plot(results, type="l", ylim=c(0,1))
par(new=T)
plot(results_p, type="l", ylim=c(0,1), col="blue") #aaの頻度を青線で表示する
}
}
wtime=function(x, y, z, a){
firsta=a
results_w=c()
for(i in 1:x){
a=firsta
results=c()
w_time=0
for(j in 1:y){
counta=0
for(k in 1:z){
if ( runif(1) < a/z ){
counta=counta+1
}
}
a=counta
if((w_time==0) && ((a==0) || (a==z))) { #もしw_timeが空で、aが0かzに固定しているとき、
w_time=j #w_timeにそのときに世代数jを入れる
}
}
results_w=append(results_w, w_time) #その集団における固定までの世代時間をresults_wに入れる
}
return(results_w)
}
drift6=function(x, y, z, a){
plot.new()
firsta=a
resultp_m=c()
for(i in 1:x){
a=firsta
results=c()
results_p=c()
for(j in 1:y){
counta=0
count_p=0
count_p_type=0
for(k in 1:z){
if ( runif(1) < a/z ){
counta=counta+1
}
}
results=append(results, a/z)
results_p=append(results_p, 2*(a/z)*(1-a/z)) #results_pに集団内のaaの頻度(count_p_type/z/2)を入れる
a=counta
}
resultp_m=append(resultp_m, results_p)
par(new=T)
plot(results, type="l", ylim=c(0,1), col="gray")
}
rmatrix=matrix(resultp_m, nrow=y, ncol=x)
hetero=c()
for(i in 1:y) {
hetero=append(hetero,mean(rmatrix[i,]))
}
par(new=T)
plot(hetero, type="l", ylim=c(0,1), col="blue")
par(new=T)
curve(0.5*(1-1/z)^x, xlim=c(0,y), ylim=c(0,1), col="red")
}