![[PukiWiki] [PukiWiki]](image/pukiwiki.png)
![[PukiWiki] [PukiWiki]](image/pukiwiki.png)
生物学の研究において、インターネットを利用したDNAデータベースへのアクセスは、必要不可欠な作業だろう。この授業では、インターネットを介してDNAデータベースシステムにアクセスし、実際に塩基配列データをダウンロードする方法を学ぶ。また、ダウンロードしたデータを用いて系統解析に挑戦する。
![[worried]](image/face/worried.png) †
†解答例
提出された課題の中から、よくできていた例:
この関数を使って、今年度前期試験に出た問題の条件で、シミュレーションをやってみよう。
集団:1万人(遺伝子の集団サイズ20000)、Hbsの遺伝子頻度、約0.1 世代数 100 地球上からマラリアが撲滅され、鎌状赤血球貧血症で死ぬことも無くなった場合
&ref(): File not found: "#12_03.jpg" at page "授業/H21/情報処理/12";
drift= function(num_repeats,num_generations,size_population, num_a_allele){
results=c()
a=num_a_allele
for(i in 1:num_repeats){
for(j in 1:num_generations){
count_a=0
for(k in 1:size_population){
if ( runif(1) < a/size_population ){
count_a=count_a+1
}
}
a=count_a
results=append(results, a/size_population)
}
a=num_a_allele
}
rmatrix=matrix(results, nrow=num_generations, ncol=num_repeats)
return(matplot(rmatrix, type="l", ylim=c(0,1)))
}
#問2-1 drift(10, 500, 200, 100)
#問2-2 drift(10, 100, 20, 10)
#前期試験 drift(3, 100, 20000, 2000) #計算には時間がかかる
![[smile]](image/face/smile.png) †
†今日の授業では、インターネットを介して情報を実際に取得して、自分の実験・解析に使うことに挑戦する。用いるのは、生物学の研究において最も頻繁に利用されるデータベースのであるDNAデータベース。リモートのサーバにデータベースが構築されており、インターネットを介してリクエストを送ることで、様々なデータを得ることができる。
今回挑戦するのは、DNAデータベースから実際にデータをダウンロードして、ローカルのコンピュータ上で加工し、系統樹を作成することだ。
現在、DNAデータベースには3つの大きなデータベースが存在する。
私自身はGenBankのEntrezという検索システムの利用に慣れているので、演習には、GenBank http://www.ncbi.nlm.nih.gov/を用いる。
まず、日本語で説明を読むために、上のリンクをクリックして、DDBJに接続してみよう。
![[smile]](image/face/smile.png) †
†それでは、早速、登録されている塩基配列情報を何か検索してみまよう。まず
GenBank http://www.ncbi.nlm.nih.gov/
に接続し、"Search"という文字の横の検索対象を"Nucleotide"にして、画面の上の方にあるテキスト入力ウィンドウに下のキーワードを入力してみよう。準備ができたら"Go"をクリックしよう。
Homo sapiens
そうするとわりとすぐに下のようなウィンドウとそれぞれの情報へのリンクが表示される。
Found 13524023 nucleotide sequences 4048555 CoreNucleotide records (通常の塩基配列データ・多くの遺伝子とその周辺領域) 8253890 EST (Expressed Sequence Tags) records (ESTライブラリで決定された塩基配列データ) 1221578 GSS (Genome Survey Sequence) records (ゲノム解析で決められた塩基配列データ)
このうち、一番上のCoreNucleotide recordをクリックしてみよう。そうすると、登録されている塩基配列がどういうものなのかを示す、サマリーという情報が表示される。 ページ右上には検索件数が表示され、1件1件はアクセッション番号にリンクがついて、リスト表示されている。アクセッション番号という名前は覚えておく方がよい。この番号は、配列につけられた固有の番号で(3大データベースで共通)、配列を研究論文で発表するときには、アクセッション番号を明記することが必須になっている。 それでは、青い文字で下線のついたリンクをクリックしてみよう。画面が変わって、登録内容が表示される。いろんな項目のことをアノテーションと呼び、登録されたデータがどの生物から得られたものかとか、遺伝子の構成、実験の条件などいろんな情報が含まれている。
#このあたりの情報のことをアノテーションと呼ぶ
LOCUS AB242157 367 bp DNA linear PLN 16-MAY-2006
DEFINITION Hibiscus tiliaceus DNA, microsatellite, clone:Ht-63.
ACCESSION AB242157
VERSION AB242157.1 GI:96775746
KEYWORDS .
SOURCE Hibiscus tiliaceus
ORGANISM Hibiscus tiliaceus
Eukaryota; Viridiplantae; Streptophyta; Embryophyta; Tracheophyta;
Spermatophyta; Magnoliophyta; eudicotyledons; core eudicotyledons;
rosids; eurosids II; Malvales; Malvaceae; Malvoideae; Hibiscus.
REFERENCE 1
AUTHORS Takayama,K., Kajita,T., Murata,J. and Tateishi,Y.
TITLE Isolation and characterization of microsatellites in the Sea
hibiscus (Hibiscus tiliaceus, Malvaceae) and related hibiscus
species
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 367)
AUTHORS Takayama,K., Kajita,T., Murata,J. and Tateishi,Y.
TITLE Direct Submission
JOURNAL Submitted (14-NOV-2005) Koji Takayama, Botanical Gardens, Graduate
School of Science, The University of Tokyo; Hakusan 3-7-1,
Bunkyo-ku, Tokyo 112-0001, Japan
(E-mail:takayama@bg.s.u-tokyo.ac.jp, Tel:81-3814-2625,
Fax:81-3814-0139)
FEATURES Location/Qualifiers
source 1..367
/organism="Hibiscus tiliaceus"
/mol_type="genomic DNA"
/db_xref="taxon:183267"
/clone="Ht-63"
/tissue_type="leaf"
repeat_region 1..367
/note="microsatellite"
/rpt_type=tandem
ORIGIN
1 taacccaaac cgccagtcca gtcttttcag cccaataccc aacacacaca ctcaacccgg
61 ctctctctct ctctatctct ctctctctca gcccactcac cctaacatag cccattcttc
121 ctttacccaa tacacacata actcactcat atacacacac acaacaaagc caacacacac
181 tctcaccctc cttcacagcc cgcaccacat actcactaac acaacccaca catatccggc
241 ctattcatac ataccaacct actcattctc acataaccca ctctcctcac aacacacaca
301 cacacacctc tcttactcaa cccatactct ctctcggccc agacctcacc tacttggccc
361 actctta
//
表示されたデータは全て、テキスト情報であることに注意しよう。
この講義の大きな目的:テキストファイル(テキスト情報)の扱いに習熟する
だったことを覚えているだろうか?DNAデータがテキスト情報で有る限り、これまで練習してきた、K2Editorなどのテキストエディタを使って編集できるということだ。また、正規表現置換・検索を行えば、自分の好きな形に加工できるということ。
さて、これで、キーワードを用いたDNAデータベースの検索は、一通りできた。あとは、Googleで検索をするときのように、キーワードを加えて絞り込むなどして、欲しい情報をデータベースから探す。
演習: なんでもいいから、自分の好きなキーワードを入れて、DNAデータベースを検索してみよう 生物の名前に限らず、人の名前でも何でも良いです
![[smile]](image/face/smile.png) †
†先ほどはキーワードでDNAの塩基配列を検索したが、BLASTを使えば塩基配列を入力して、同じような配列を持つ登録データがあるかどうかを調べることもできる。DNA塩基配列における"Googleサーチ"のようなものだ。BLASTNをクリックして、BLASTの検索画面を表示させててみよう。
(GenBankのトップページからリンクを辿って、BLASTのページに入り、"Nucleotide-nucleotide BLAST (blastn)"をクリックすることでも入れる)
次に、真ん中あたりの「Choose Search Set」で、Others(nr etc):をクリックしておこう。
&ref(./#12_1.jpg)
ctctacaagt attgtaattt taagagtctt tttactccaa agaaatcccc tttttttttg
それでは、検索ウィンドウに上の60ベースの塩基配列を入れ、![]() というボタンをクリックしてみよう。他にもいろいろとオプションの設定はあるが、無視してかまわない。
というボタンをクリックしてみよう。他にもいろいろとオプションの設定はあるが、無視してかまわない。
BLAST!をクリックすると次の画面が表示されるが、検索にはしばらく時間がかかる。画面には経過時間が表示される。 検索が終わると、検索が表示される。画面の上の方には、結果がグラフィックで表示され、、画面の下の方には、説明がテキストで書かれている。
この画面では、先ほど入力した配列をデータベースサーチして、よく似た配列ほど、上から順に高いスコアで表示される。実は、上の60塩基の配列は、Dipterocarpus kerrii というフタバガキ科の植物からとってきたものだが、検索結果の最初の3つは、全く同一スコアで、その中の一つはDipterocarpus kerii。この検索の結果、先ほどの配列は、Dipterocarpusの葉緑体DNAにあるmatKという遺伝子の配列に含まれているものに非常に近いということが分かる。
実験で得られた遺伝子の塩基配列から、似た遺伝子を探して働きを推測するときに、BLASTサーチは非常に有効。
演習: 50塩基ぐらいの配列を自分で考えて作って、BLASTサーチしてみよう。高スコアで何かの遺伝子と一致するか?
![[smile]](image/face/smile.png) †
†先に行ったキーワード検索では、表示されたサマリーから遺伝子の情報を表示させただった。でも、生物学の研究では、複数の塩基配列情報を、1つのファイルにまとめて保存したいことがよくある。
例えば、皆さんの卒業研究では、次のような場面でDNAデータベースからデータをダウンロードすることになる。
etc...
研究テーマにもよるが、生物学のほとんどの研究分野でDNAデータベースからのデータを取得する場面が出てくる。
それでは、複数の配列データを一括ダウンロードしてみよう。先ほどはキーワードで検索を行ったが、今度はアクセッション番号で検索してみよう。これから先、実際に研究を行うときに、ある論文で発表されている塩基配列をDNAデータバンクから得ようとすると、アクセッション番号を使ってダウンロードすることが多い。
下の囲みの中には、ヒト、ゴリラ、チンパンジーのミトコンドリアDNAの全配列を研究した論文(日本語要約、系統樹)から、日本人、フランス人、アフリカ人(Lisongo)、チンパンジー、ゴリラのアクセッション番号が挙げられている。
AF346989,AF346981,AF346994,D38113,D38114
GenBankやDDBJのgentryというシステムで検索するときは、アクセッション番号をコンマで区切って検索欄に入力すると、対応する配列だけが表示される。では、上の囲みの中の文字列をコピーして、GenBankの検索欄にペーストし、Nucleotideを検索してみよう。
CoreNucleotideの左の5という数字をクリックすると、5つの遺伝子のサマリーが表示されただろうか? え?日本人とか、フランス人とかいう情報がサマリーに表示されていないって?... そのとおり。サマリー情報には私たちが使いたい情報が載っているとは限らないので、アクセッション番号がどの遺伝子に対応しているかは、それぞれの詳細情報を見ないと分からない場合がある。それでは不便なので、アクセッション番号と、自分の使いたい情報の対応表を作りたいところだが、これまで学習してきた正規表現検索・置換を使えば、簡単に対応表をつくることができる。これについては、後で時間があったら、演習をやってみるよてい。
さて、自分の指定したアクセッション番号を持つ5つの配列が画面に表示された。次はこれを一括ダウンロードしよう。ダウンロードするには、画面の上のほうにあるSend toと書かれたプルダウンメニューをクリックし、Fileを選ぶ。そうすると、ブラウザがファイルを保存するかどうか聞いてくるので、保存する。
私が今使っているシステムでは、 sequences.fasta という名前で配列情報が保存された。これをテキストエディタ(K2Editorなど)で開いてみると、
>gi|13272920|gb|AF346989.1| Homo sapiens mitochondrion, complete genome GATCACAGGTCTATCACCCTATTAACCACTCACGGGAGCTCTCCATGCATTTGGTATTTTCGTCTGGGGG GTGTGCACGCGATAGCATTGCGAGACGCTGGAGCCGGAGCACCCTATGTCGCAGTATCTGTCTTTGATTC .......................
塩基配列情報が入っていた。
なお、保存されたファイルをK2Editorで開いたとき、下のように塩基配列じゃなくてサマリーがテキストファイルとして保存されている人がいたかもしれない。これは、よくやる間違いの一つで、プルダウンメニューの横の方にかかれているSummaryの所を、FASTA形式に変更し忘れたためにおきたこと。上の説明を読んで、もう一度やってみよう。
1: AF346989 Homo sapiens mitochondrion, complete genome gi|13272920|gb|AF346989.1|[13272920] 2: AF346981 Homo sapiens mitochondrion, complete genome gi|13272808|gb|AF346981.1|[13272808]
今ダウンロードした塩基配列はミトコンドリアDNAの全長なので、およそ1万6千ベースある。非常に長いため、テキストエディタで表示させても、データの区切りがどこにあるか分かりにくいですね。ともかく、今の操作で5つのDNAデータがダウンロードできた。この後のアラインメントなどの操作は、DNAのマルチプルシークエンスアラインメント解析専用のソフトウェアを使って行いる。
FASTA形式は、複数の塩基配列をタを並べて扱うときに用いる形式の1つ。FASTA形式は非常にシンプルなデータ形式をしており。今では、GenBankのBLAST検索や、様々な塩基配列解析ソフトウェアで広く使われている(FASTA形式の詳しい説明はこちら)。
簡単に説明すると、
>配列名などの情報 塩基配列またはアミノ酸配列
という構造になっている。下の囲みの中の配列は、ダウンロードした配列から、テキストエディタ(K2Editor)を使って、私が適当に作ったFASTA形式ですが、このままで十分、アラインメント解析ソフトで解析することができる。
>Japanese TTGACCGCTCTGAGCTAAACCTAGCCCCAAACCCACTCCACCTTACTACCAGACAACCTTAGCCAAACCATTTACCCAAATAAAGT ATAGGCGATAGAAATTGAAACCTGGCGCAATAGATATAGTACCGCAAGGGAAAGATGAAAAATTATAACCAAGCA >French CTTGACCGCTCTGAGCTAAACCTAGCCCCAAACCCACTCCACCTTACTACCAGACAACCTTAGCCAAACCATTTACCCAAATAAAGT ATAGGCGATAGAAATTGAAACCTGGCGCAATAGATATAGTACCGCAAGGGAAAGATGAAAAATTATAACCAAGC >African TGACCGCTCTGAGCTAAACCTAGCCCCAAACCCACTCCACCTTACTACCAGACAACCTTAGCCAAACCATTTACCCAAATAAAGTAT AGGCGATAGAAATTGAAACCTGGCGCAATAGATATAGTACCGCAAGGGAAAGATGAAAAATTATAACCAAGCAT >Chimpansee ACTCTGAGCCAAACCTAGCCCCAAACCCCCTCCACCCTACTACCAAACAACCTTAACCAAACCATTTACCCAAATAAAGTATAGGCGA TAGAAATTGTAAACCGGCGCAATAGACATAGTACCGCAAGGGAAAGATGAAAAATTATACCCAAGCATAATA >Gorilla GCTCTGAGCAAAACCTAGCCCCAAACCCACCCCACATTACTACCAAACAACTTTAATCAAACCATTTACCCAAATAAAGTATAGGCGA TAGAAATTGTAAATCGGCGCAATAGATATAGTACCGCAAGGGAAAGATGAAAAAATATAACCAAGCACGACAC
塩基配列の区切りに >生物名(改行) を入れれば、いろんなソフトウェアで解析ができるんだから、K2Editorに慣れた皆さんにとっては、とても親しみやすい形式だろう。
![[smile]](image/face/smile.png) †
†アラインメントというのは、複数の塩基配列情報やアミノ酸の配列情報を整列させることだ。塩基配列情報を扱う上でとても重要な言葉なので、覚えておおこう。例えば、
cytochrome b遺伝子: ヒト ..attaaccccctaataaaattaattaaccactcattcatcgacctccccaccc... ゴリラ atgacccctatacgcaaaactaacccactagcaaaactaattaaccactcattc...
という2つの配列はアラインメントされていない。ヒトとゴリラという異なる種から得られた配列だけれど、同じ遺伝子なので、きっと相同な領域はあるに違いない。しかし、こういう並べ方をすると、塩基配列のどの位置がどの位置に対応しているのか分からない。これをアラインメントすると、
cytochrome b遺伝子のアラインメント: ヒト atgaccccaatacgcaaaattaaccccctaataaaattaattaaccactcattcatcgacctccccaccccatc ゴリラ atgacccctatacgcaaaactaacccactagcaaaactaattaaccactcattcattgacctccctaccccgtc 塩基置換 * * * ** * * * *
となり、サイト(塩基配列上の塩基一つ一つの位置のこと)ごとに対応関係をとることができるし、どのサイトで塩基置換が生じているのかが、一目でわかる。
異なる生物から得られた塩基配列を複数並べて、構造上の対応関係を見たり、系統樹を作成する場合は、用いる塩基配列がアラインメントされていることが必須だ。そこで、皆さんのコンピュータに、代表的なアラインメントソフトウェアである、ClustalXをダウンロードして、インストールしよう。
下のリンクのいずれかをクリックしてみよう。下の方をクリックした場合は、clustalx1.83.zipというファイルをリストから探して、ダウンロードしよう。
それでは、下のサンプルファイルをダウンロードしてみよう。
このファイルには先ほどGenBankで検索したヒトのミトコンドリアDNAの配列の一部がFASTA形式で保存されている。ダウンロードされたファイルは、デスクトップに(デスクトップに無ければマイドキュメントに)入っているはず。
ClustalXの画面に移動し、example1.fasta を読み込む。
操作: Alignmentメニュー / DoCompleteAlignment
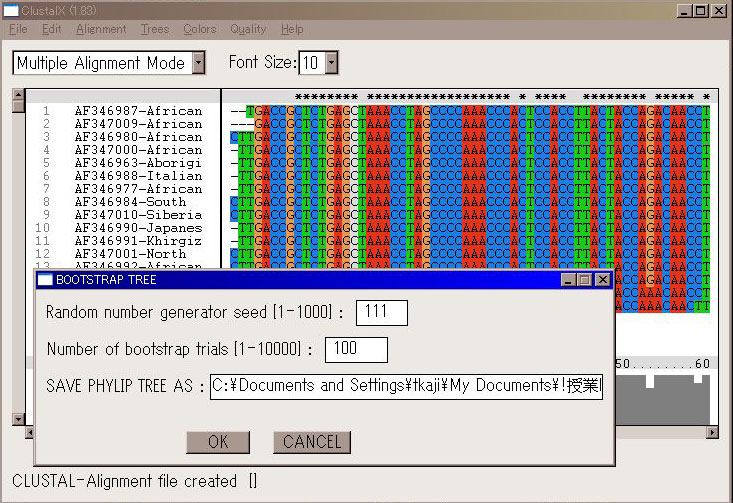
操作: Treeメニュー / Bootstrap N-J Tree : 2番目のカラム 1000 -> 100 結果はもとのファイルと同じ場所の「元のファイル名.phb」 という名前のファイルに保存されている
先ほどClustalXで作成した系統樹を画面に表示させてみよう。
ここまでできれば、DNAデータバンクからデータをダウンロードして、系統樹を描くことに成功ということ。
*第12回授業・基本課題 **氏名: **課題への回答 -今日の授業の進み方は?(はやい、丁度いい、おそい) --回答: -今日の授業の難しさはどう感じましたか(簡単 丁度いい 難しい): --回答: -難しいと答えた人は、特にどの点が難しかったですか?: --回答: -今日の授業は(分かった 半分ぐらいは分かった 分からなかった): --回答: -分からないと答えた人は、特にどの点が分からなかったですか?: --回答: -今日の講義で分からなかった用語があったら挙げてください: --回答: -授業に関する要望・質問があったらなんでもどうぞ: --回答: -課題2の答え --問1: --問2: ---Accession No.: ---Description: --問3: --問4:
D88776,D88085,D88087,D88088,D88089,D88090,D88092,D88093,D88094,D88096,D88097,D88098,D88099,D88100,D88101 上アクセッション番号の配列15個を、GenBankで検索しなさい。 検索結果をFASTA形式でダウンロードして、ClustalXでアラインメントし、NJ法で系統樹を作成して、 NJPlotで系統樹を表示しなさい。外群にはD88101(エゾシオガマ)を指定しなさい。可能ならば BootStrap解析を1,000回(あるいは100回)行いなさい。
D88776 GASSAN 月山 D88085 UNALASKA ウナラスカ D88087 DAISETSU 大雪山 D88088 IWATE 岩手山 D88089 RISHIRI 利尻 D88090 POROSHIRI 幌尻岳 D88092 AKITAKOMA 秋田駒 D88093 HAYACHINE 早池峰山 D88094 IIDE 飯豊山 D88096 ONTAKE 御岳 D88097 KISOKOMA 木曽駒 D88098 YAYSUGATAKE 八ヶ岳 D88099 KITADAKE 北岳 D88100 ARAKAWA 荒川岳 D88101 yezoensis 注:地名は全て高山帯のある山または地域。