生物学セミナー †
授業情報 †
2007年度 授業コード:S4302101 科目コード:S43021
前期・ 火5 単位数 :2 専門科目(生物学科) 教室 5月12日以降は407号室
授業科目名: 生物学セミナー (Seminar in General Biology)
目的・目標 †
生物学入門。大学でおこなわれている高度な生物学に早期に触れさせ、学問への動機づけを行う。
授業計画・授業内容 †
1−15 前半は、オムニバス形式で、各研究分野の教官の研究内容の概略を学ぶ。後半は2,3人ずつに分かれ、1人の教官が生物学各分野への入門的な基礎文献の購読ならびに生物の観察、実験手法等の指導を行う。
講義予定(進行状況に応じて変更するので注意!) †
評価方法・基準 †
出席を重視。後半の小人数教育では、教官から与えられる課題への積極的な取組みが求められる。
初回:6月11日 †
学生2名とセミナー実施内容を相談。「植物観察実験」、「DNAデータの解析実験」、「研究論文の読み方」などの選択肢から、学生の希望により、「漂着種子を用いた海流散布植物の漂着ルートの解明」を選択。
目的 †
- 房総半島に漂着するモダマ(Entada phaseoloides)種子を採集し、葉緑体ハプロタイプを決定することにより、台湾、琉球地域等、どの集団に由来するものかを推定する。
- フィールド調査:7月8日(日)
- 調査内容:
- 房総半島に漂着する熱帯植物の種子(とくにモダマ)の採集
- ハマナタマメの採集
予備調査 †
- 以下の文献を精読して、海流散布種子のテーマについて考えておく
7月8日現地調査: †
採集した漂着種子 †
発芽実験 †
- 房総半島で採集したハマナタマメの種子8個、種皮に傷をつけて吸水
DNA抽出 †
| 1 | ハマナタマメIga | N34 56'28.3", E 139 48'57.1" | 4 |
| 2 | グンバイヒルガオ芽生え | 〃 | 4 |
| 3 | グンバイヒルガオ種子 | N 35.00'32.8" , E 139 58'46.5" | 4 |
| 4 | グンバイヒルガオ種子 | N 35.00'32.8" , E 139 58'46.5" | 3 |
| 5 | グンバイヒルガオ種子 | N 35.00'32.8" , E 139 58'46.5" | 7 |
| 6 | ハマナタマメ | | 3 |
| 7 | ハマエンドウ1 | | 3 |
| 8 | ハマエンドウ2 | | 10 |
| 9 | ハマエンドウ3 | | 7 |
| 10 | ハマエンドウ4 | | 10 |
| 11 | E.reticulata (seed) | | 1 |
| 12 | E.grandulosa (seed) | | 1 |
塩基配列解析の準備 †
塩基配列を決定する実験を行う前に、手持ちのPCR(or sequence) プライマーが使えるかどうか、あるいは、どのような組み合わせで使えば効率よく実験が行えるかを、次のような手順で調べておく必要がある。
- BioEditを次のサイトからダウンロードしてインストール http://www.mbio.ncsu.edu/BioEdit/bioedit.html
- File >> New Sequenceから新規シーケンス編集画面に入るか、近縁種のシーケンスをNCBI http://www.ncbi.nlm.nih.gov/ からFasta形式でダウンロードして、BioEditで開く。
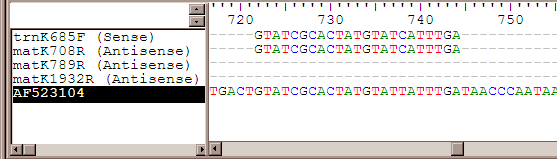
- プライマー名とプライマーシーケンスのリストをタブ区切りで準備しておく(下例: 空白部分はタブ)
trnK685F GTATCGCACTATGTATCATTTGA
matK708R TCAAATGATACATAGTGCGATAC
matK789R TAGGAAGTCCTGNTGGCGAGATC
matK4L CTTCGCTACTGGGTGAAAGATG
matK4R CATCTTTCACCCAGTATCGAAG
matK1777L TTCAGTGGTACGDAGTCAAATG
matK1777R CATTTGACTHCGTACCACTGAA
matK1932R CAGACCGGCTTACTAATGGG
- BioEditで比較したい配列を選択し、「Sequence >> PCR Primers / oligos >> Map antisense oligos」を選ぶ。開いた小さいウィンドウに、上のタブ区切りリストをペースト。max mismatchでいくつまでミスマッチを許すかを指定する。
- プライマーリストのうちマッチmax mismatchの範囲でマッチするものだけが、選択した配列上に示される。配列を比較することで、そのプライマーが使用可能かどうかの見当をつける。
- 適当なプライマーが見つからなかった場合、Primer3 http://frodo.wi.mit.edu/cgi-bin/primer3/primer3.cgi を用いて、新規プライマーとして適当な配列を検索する。その際、複数の配列から作成したコンセンサス配列を用いる方が良い。
- Entadaの既存配列からtrnKを増幅する最適プライマーの位置を検索した結果:
- Cycle Sequencing
| A | A4 12Entgra | A8 12Entgra | B2 12Entgra | B4 12Entgra |
| 685F | matK4R | matK1777F | matK2R | trnL-c | trnL-d | trnL-e | trnL-f |
![[PukiWiki] [PukiWiki]](image/pukiwiki.png)